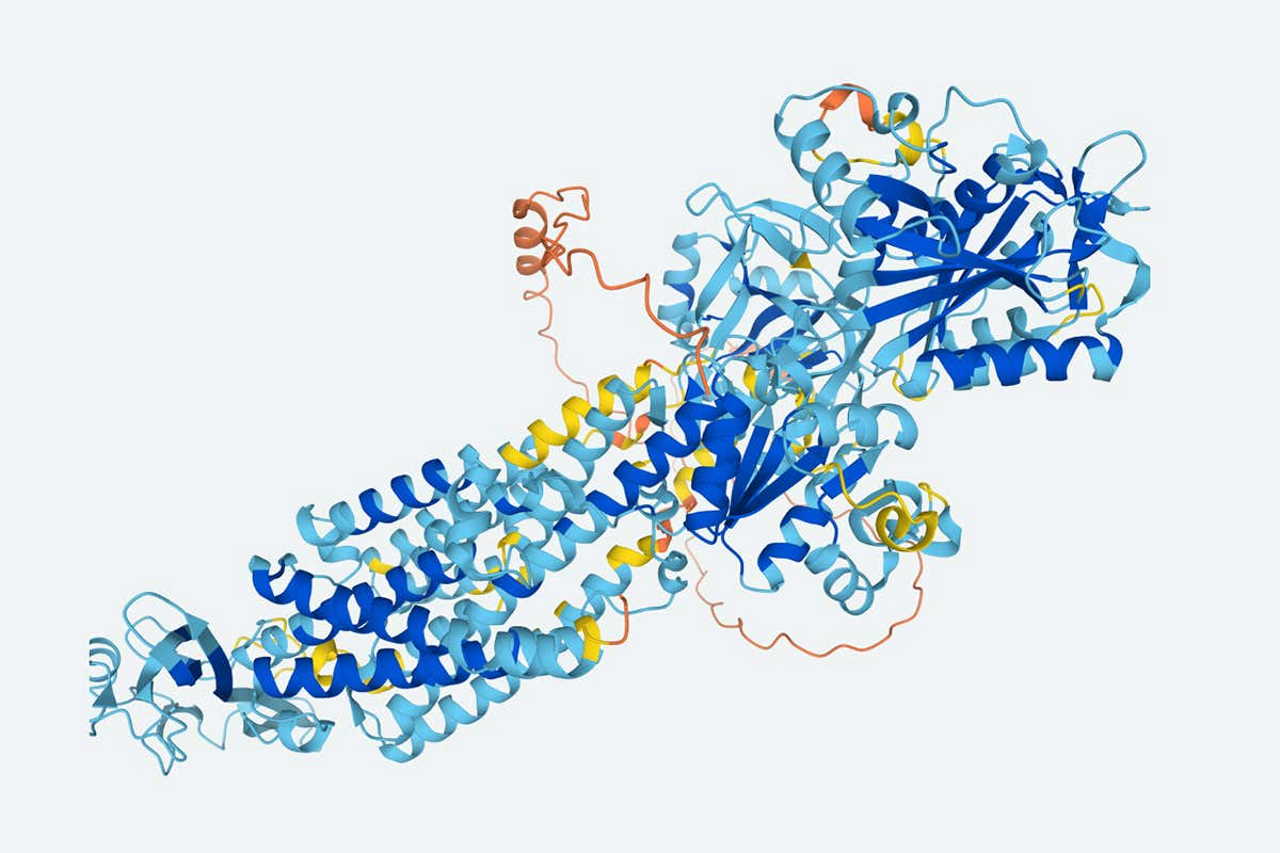
Розробникам DeepMind вдалося створити каталог структур близько 20 тисяч білків, що кодуються людським геномом. Це 98,5 відсотка протеома людини. Разом з ними алгоритм AlphaFold надав у відкритий доступ до структур білків ще 20 організмів — від плодової мушки до кишкової палички. Отримані результати і сам код AlphaFold вчені надали у відкритий доступ, а стаття про роботу доступна у Nature.
Навіщо ШІ розгортав людські білки?
Експериментальне визначення структури білків вимагає подолання багатьох труднощів, що віднімають багато часу: білку для дослідження має бути достатня кількість, причому очищеного та зібраного у відповідних умовах. Будь-який з цих етапів може виявитися надскладним, а перешкодою може стати розмір білка, наявність трансмембранних областей або схильність до конформаційних змін структури. Тож всеосяжне структурне дослідження протеома залишається непростим завданням — у нас немає єдиного методу. Поки зусиллями окремих лабораторій і спеціальних ініціатив (як-то проєкт «Протеом людини») ми маємо більше 50 000 структур білків людини у банку тривимірних структурних даних білків і нуклеїнових кислот.
Однак, поки лише 17 відсотків мають визначену та експериментально підтверджену структуру. Тому прогнозування структури білка сприяє скороченню цієї прогалини, оскільки дає можливість швидко і масштабно отримувати гіпотези щодо структури білків. Тому з'явився всесвітній експеримент КАСП — Критичний Аналіз методів для Структурних Передбачень білків. З 1994 року десятки міжнародних команд дослідників раз на два роки змагаються у тому, чий метод моделювання найкраще передбачить ще неопубліковану, однак відому організаторам, майбутню форму 100 амінокислотних послідовностей. DeepMind на “змаганнях” з’явилися 2018 року і тоді ж посіли перше місце з алгоритмом AlphaFold, який оцінили у 90 балів зі 100. Минулого року цей алгоритм передбачив структури десятків білків за кілька днів з похибкою лише в 0.16 нанометра — розміром з атом.
У чому проблема білків?
Перш ніж білок набуває своєї тривимірної форми — згортається — він перебирає величезну кількість послідовностей амінокислот. Причому робить він це спонтанно, бо повний перебір можливих конформацій зайняв би 1080 років. Однак, саме від його форми залежить функція, яку він виконуватиме, тому з 1970-х років, коли цю закономірність виявили, над біохіміками нависла так звана «проблема згортання білків». Швидкий, дешевий і точний спосіб отримання таких структур сильно б спростив життя молекулярним біологам і медикам, але поки їм доступні лише складні експериментальні методи кріоелектронної мікроскопії, ядерного магнітного резонансу або рентгенівської кристалографії, що вимагають місяців та років часу, складних умов та часто кількох спроб і помилок.
Що зробив AlphaFold?
Структура AlphaFold містить два алгоритми, які обмінюються результатами між собою і таким чином уточнюють його. Один з цих алгоритмів шукає білкові послідовності, які є еволюційними «родичами» розглянутого із загальнодоступного сховища послідовностей і структур, і визначає, як їхні послідовності згортаються. Навіть якщо ми не знаємо повну структуру будь-якого з цих порівнюваних білків, вони надають важливі обмеження, повідомляючи, наприклад, про окремі частини структури, які впливають на згортання. Другий алгоритм працює паралельно і розбиває отриману першим послідовність амінокислот на дрібніші фрагменти, структуру кожного з яких намагається сумістити із загальною. Так алгоритму вдалося розв'язати структуру 98,5 відсотка білків людини, а всього близько 20 тисяч послідовностей. Причому розробники надали доступ і до протеома 20 інших організмів, як-то плодова мушка, миша, дріжджі і кишкова паличка, що в цілому становить понад 350 тисяч білкових структур.
За їхніми словами, вони планують розширити «каталог» ще принаймні сотнею мільйонів білків, щоб створити «білковий альманах світу». У своїй статті вони висвітлили кілька структурних гіпотез, для яких досі взагалі не було припущень щодо структури. Наприклад, глюкозо-6-фосфатаза, що є мембранним ферментом, який бере участь у заключному етапі синтезу глюкози та відіграє ключову роль в регуляції рівня цукру у крові. За оцінкою, точність отриманої структури сягає 95,5 (зі ста), що може значно допомогти у дослідженнях. Також із точністю 95.9 вдалося передбачити структуру діагліцеролу-О-ацілтрансферази 2 — білку, який відповідає за накопичення жиру, для якого вдалося навіть спостерігати специфічні взаємодії. Меншої точності (81,7) вдалося досягти у структурі білку WFS1, який пов'язаний з нейродегенеративним захворюванням синдромом Вольфрама, що характеризується раннім початком діабету, поступовою втратою зору і слуху та ранньою смертю. Однак, з огляду на низьку впевненість вчених у передбаченні алгоритму, вони вирішили ідентифікувати окремі ділянки, унікальні для цієї структури.
Наскільки можна довіряти алгоритму?
Щоб прогнози були не просто спробою подивитися, як алгоритми можуть розв'язувати такі завдання, а і були практично корисними, вони повинні мати визначену міру достовірності. Саме тому розробники DeepMind не намагалися передбачити структуру кожного білка в геномі людини — деякі просто занадто великі, щоб з ними було зручно працювати. Тож вони встановили граничний розмір на рівні 2700 амінокислот. Втім, все ж більшість білків підходять під ці параметри, тому їх вдалося передбачити таку кількість. Метрику достовірності розробники оцінили за шкалою від 0 до 100. Підхід оцінює, наскільки добре прогноз буде узгоджений з експериментальною структурою на основі тесту локальної різниці відстаней (The Local Distance Difference Test, lDDT). Так у діапазон максимальної точності потрапило 35,7 відсотка всіх спрогнозованих структур, 58 відсотків передбачені з упевненістю більше 70, що є нижнім граничним значенням і відповідає в цілому правильному прогнозу ланцюжка, а 1 290 білків містять понад 200 послідовностей, щодо прогнозів яких не можна говорити точно. Загалом алгоритм впевнений у прогнозах близько 60 відсотків структур, а з високим ступенем впевненості — трохи більше третини. Очевидно, що поки це не повний набір білків людини, однак це набагато більше за відомі 17 відсотків на сьогодні.